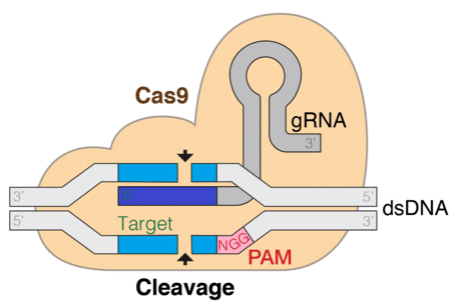
La tecnología CRISPR, conocida por su capacidad transformadora en la edición genética, continúa avanzando con investigaciones que buscan mejorar su eficacia y precisión. Un equipo de investigadores ha adoptado un enfoque innovador utilizando modelos de lenguaje de gran escala (LLMs) para predecir la eficiencia del ARN guía (gRNA), una pieza crucial en el mecanismo de CRISPR-Cas9.
El ARN, que es transcrito a partir de secuencias de ADN, se pliega en estructuras tridimensionales intricadas y desempeña un rol esencial en la edición genética. Para abordar la predicción computacional de la eficiencia y especificidad del gRNA, los investigadores han aprovechado los modelos de lenguaje previamente entrenados con vastas cantidades de secuencias biológicas.
En esta investigación se empleó un modelo genómico preentrenado, conocido como DNABERT, para predecir la eficiencia del gRNA. Este modelo trata cada secuencia de gRNA como una oración, aplicando técnicas de regresión similares al análisis de sentimientos en el lenguaje humano. Utilizando Amazon SageMaker y la biblioteca PEFT de Hugging Face, los investigadores ajustaron eficientemente el modelo DNABERT para procesar datos de ARN y determinar la puntuación de eficiencia basada en condiciones experimentales.
Los resultados obtenidos demostraron que ajustar todos los parámetros de un LLM convencional es costoso en términos computacionales. Por ello, se utilizó una técnica conocida como LoRA (Adaptación de Baja Rango). Esta técnica permite mantener fijos la mayor parte de los pesos del modelo mientras se entrenan capas adicionales, reduciendo significativamente el número total de parámetros y los requisitos de memoria de la GPU.
Para evaluar el rendimiento del modelo, se emplearon métricas como el Error Cuadrático Medio (MSE), Error Medio Absoluto (MAE) y la Raíz del Error Cuadrático Medio (RMSE). Los experimentos mostraron que el uso de LoRA con un rango de 8 proporcionó resultados competitivos, con un RMSE de 11.933 y un MSE de 142.397, lo cual es cercano al modelo estándar CRISPRon. Sin embargo, con un rango de 16, se observó un ligero empeoramiento en las métricas, sugiriendo un posible sobreajuste.
La comparación final indica que aunque LoRA mostró un rendimiento ligeramente inferior a CRISPRon, sus beneficios en términos de eficiencia computacional y reducción de parámetros lo hacen una opción prometedora con potencial para superar a los modelos existentes mediante una optimización de hiperparámetros más exhaustiva.
En resumen, esta investigación no solo destaca el potencial de los LLMs en biología computacional, sino que también proporciona un camino eficiente para mejorar las tecnologías actuales de edición genética mediante la predicción precisa de la eficiencia del gRNA. Estas innovaciones son cruciales para el avance en el tratamiento de enfermedades y la comprensión del genoma humano.